インストール方法
APBS(Adaptive Poisson-Boltzmann Solver)は、タンパク質の静電ポテンシャル(表面電荷)を計算するソフトウェアです。オープンソースであり、Fink を用いてインストールできます。APBS は PyMOL から簡単に操作できるのでお勧めです。ただし、MacPyMOL からは操作できませんのでオープンソースの PyMOL をインストールしていない場合は、Fink を用いて APBS といっしょにインストールしましょう。ターミナルを立ち上げて、以下のコマンドを実行します。APBS と PyMOL が依存しているソフトも自動的にインストールされます。インストール開始直後に何回か一時停止し、”dependencies” について聞いてきますが、基本的にリターンのみで大丈夫なはずです。
fink install apbs pymol-py27
静電ポテンシャル表示の簡単なチュートリアル
APBS はファイルをいくつか作成するので、新しいフォルダの中で作業すると良いでしょう。ここでは、デスクトップに新しいフォルダを作成します。新しいフォルダの場所はホームディレクトリの中であればどこでも大丈夫ですが、ディレクトリのパスにスペースがないことを確認してください。スペースがあると、APBS は正常に動作しません。フォルダを作成するには、マウスのポインタをデスクトップに移動させ、右クリック(または control キー + 左クリック)でコンテキスト・メニューを出し、一番上の「新規フォルダ」を選びます。フォルダの名前は “名称未設定フォルダ” から “surface_potential” に変えます。次に、フォルダを Finder で開き(アイコンをダブルクリック)、ツールバーの「ターミナルを開く」をクリックしてターミナルを立ち上げます。まず以下のコマンドで PyMOL を立ち上げましょう。
/sw/bin/pymol
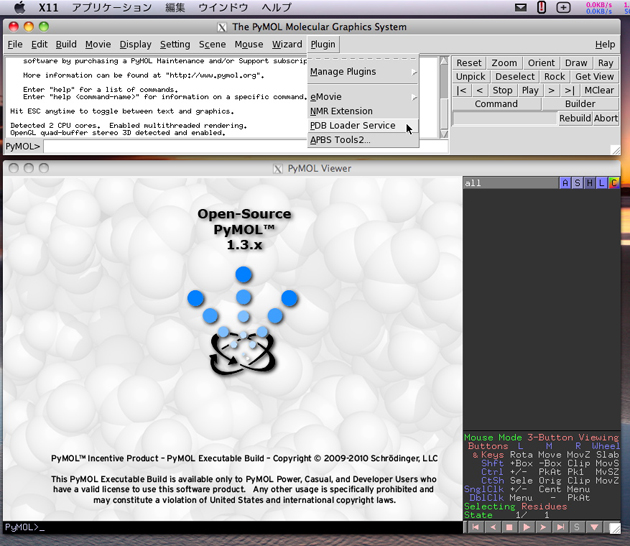
次に PDB を表示します。ここでは、カプシドタンパク質(ウイルスゲノムを取り囲む殻を構成するタンパク質)と RNA アプタマーの複合体を用います。PyMOL のメニューの Plugin > PDB Loader Service を選択します。
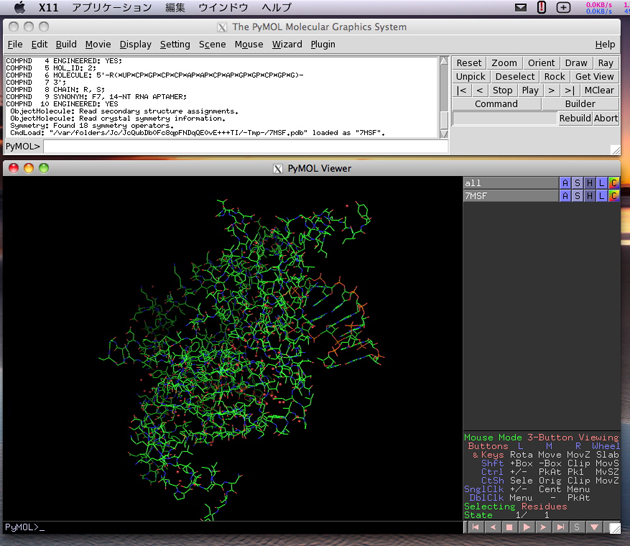
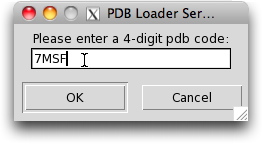
新しいウインドウが開きますので、”7MSF” と入力し、OK をクリックします。7MSF は Protein Data Bank に登録されているカプシドタンパク質・RNA 複合体の ID です。Protein Data Bank のサーバーからダウンロードするので、インターネットにつながっている必要があります。
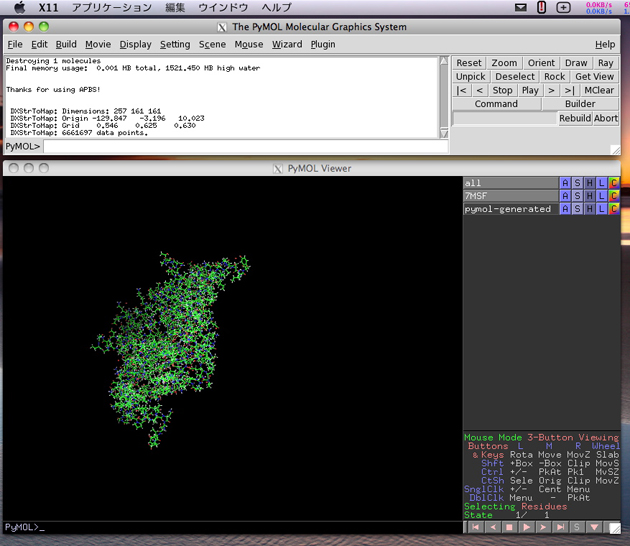
ダウンロードに成功すると、PyMOL のメインウインドウに複合体の座標が表示されます。
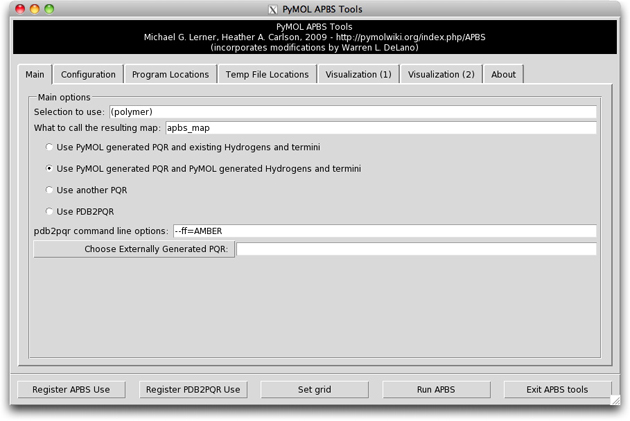
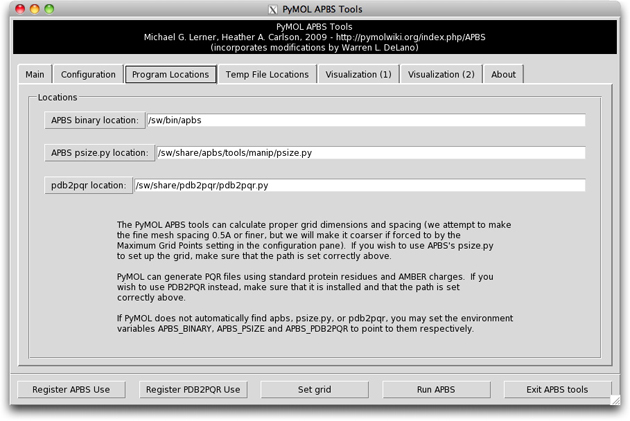
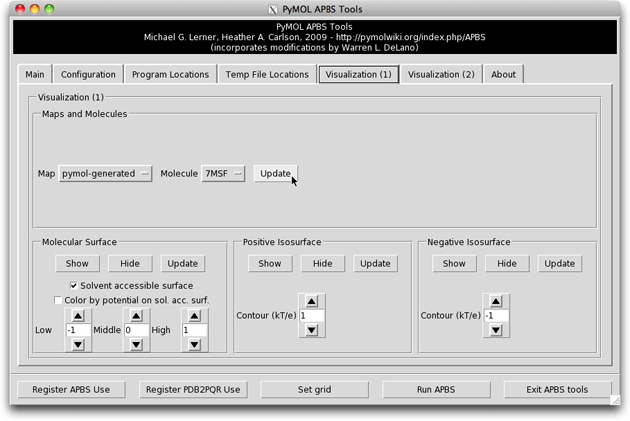
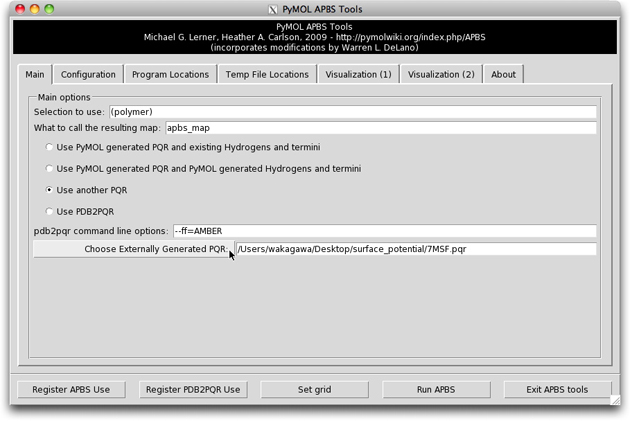
PyMOL のメニューの Plugin > APBS Tools … を選択します。Main と APBS Location タブの中の設定は以下の通りになっているはずです。
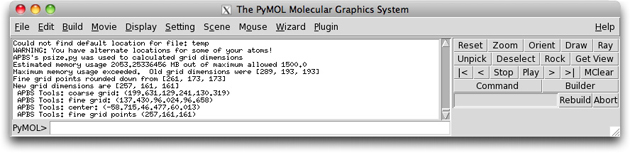
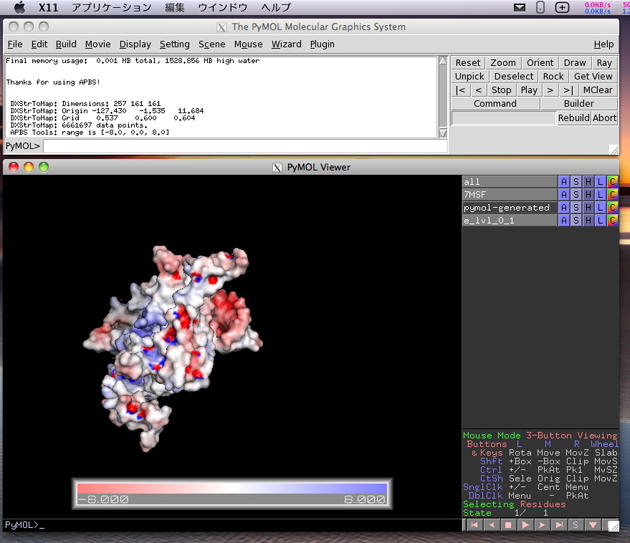
続いて、APBS ウインドウの Set grid ボタンをクリックします。問題がなければ、PyMOL の上のウインドウ内に以下のログが表示されます。
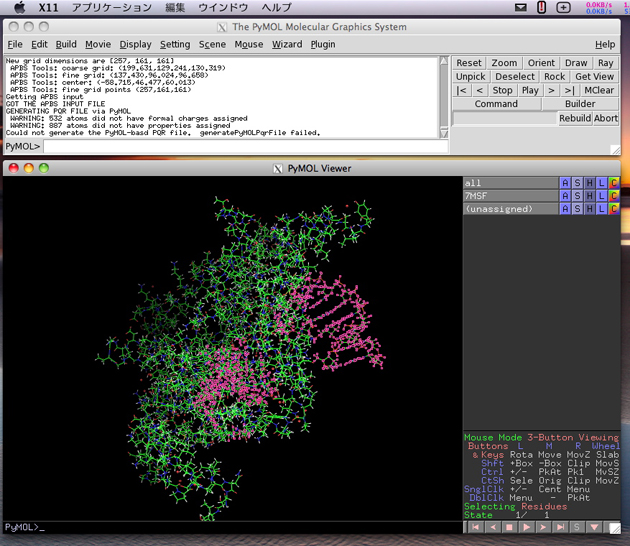
次に APBS ウインドウの Run APBS ボタンをクリックし、静電ポテンシャルを計算します。しばらくすると、「一部の原子について静電ポテンシャルを計算できませんでした」という内容のメッセージが表示されます。
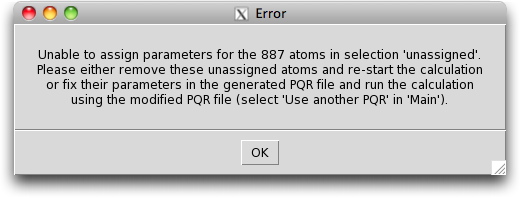
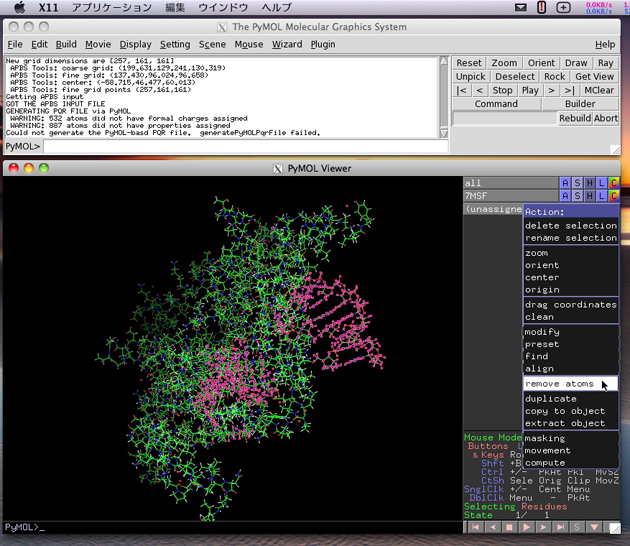
静電ポテンシャルを計算できない原子は、”unassigned” という名前で選択されます。この中に RNA が含まれています。これらを削除して残りの原子の静電ポテンシャルを計算する場合は、(unassigned) の Action ボタンをクリックし、remove atoms を選択します。
再度、”Set grid” と “Run APBS” を実行すると以下のようになります。
APBS ウインドウの中は、自動的に Visualization (1) のタブに切り替わります。念のため、”Maps and Molecules” の Update ボタンをクリックします。
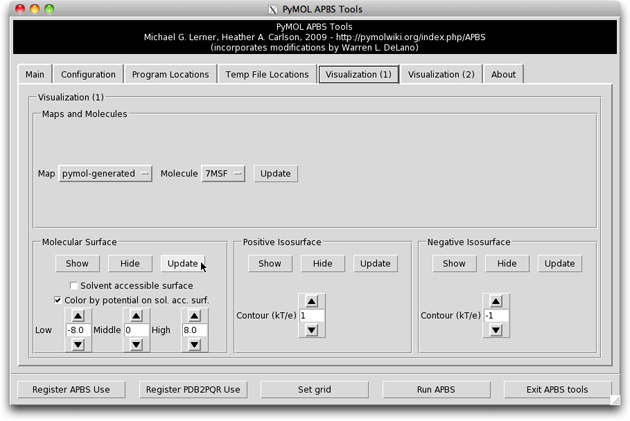
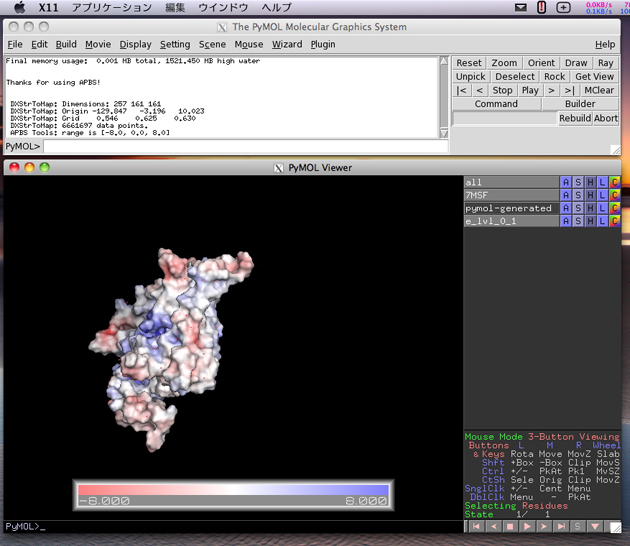
次に、”Molecular surface” の中の静電ポテンシャルの範囲を設定し(ここでは -8 kT から +8 kT)、”Color by potential on sol. acc. surf.” にチェックを入れます。すぐ上の Update ボタンをクリックするとタンパク質の表面が表示され、計算された電荷分布に従ってカラーリングされます。
上記では、一部の原子について静電ポテンシャルを計算できませんでしたが、これは PyMOL が作成した PQR ファイルと APBS との相性が良くないからです。表面電荷を表示したい座標に、通常の 20 種類のアミノ酸以外のもの(核酸、低分子化合物、金属イオンなど)が含まれている場合は、pdb2pqr というソフトウェアで事前に PQR ファイルを作成し、それを APBS に読み込ませてから表面電荷を計算すると良いでしょう。pdb2pqr は、APBS をインストールしたときに自動的にインストールされますので、すぐに作業を始められます。
まず PyMOL を一旦終了し、surface_potential のフォルダの中で PyMOL を再起動します。そして 7MSF の座標を上記の手順でもう一度表示させます。次に、PyMOL のメニューで File > Save Molecule … を選択し、座標を7MSF.pdb という名前で surface_potential のフォルダの中に保存します。PyMOL を終了し、ターミナルで以下のコマンドを実行して PQR ファイルを作成します。
pdb2pqr --ff=amber 7MSF.pdb 7MSF.pqr
今度は PyMOL の立ち上げと同時に 7MSF.pdb を開きます。
/sw/bin/pymol 7MSF.pdb
次にAPBS ウインドウを開き、Main のタブの中の “Use another PQR” をチェックし、”Choose Externally Generated PQR” のボタンをクリックして、7MSF.pqr を選びます。
あとは “Set grid” と “Run APBS” を実行すれば、複合体全体の表面電荷が計算されます。
PyMOL で、File > Save Session で保存したセッションは、MacPyMOL で開くことが可能です。
最終更新日:2012年11月18日